Neuentwicklung von Biokatalysatoren
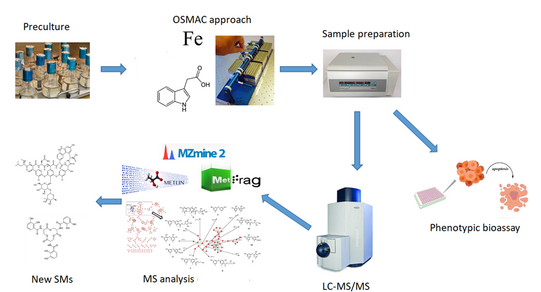
Zahlreiche Enzyme und Sekundärmetabolite schlummern in Mikroorganismen. Sie können durch traditionelle Screening-Ansätze nur schwer entdeckt werden. Die Bioprozesstechnik arbeitet daher mit innovativen Methoden wie dem Genome Mining oder Sekundärmetabolit-Screenings nach dem "One strain many compounds"-Ansatz (OSMAC).
Metabolomics
Gegenwärtig besteht ein enormer Bedarf an neuartigen Wirkstoffen für die Behandlung von Krankheiten und insbesondere zur Bekämpfung multiresistenter mikrobieller Krankheitserreger. Mikrobielle Naturstoffe, vor allem Sekundärmetaboliten, spielten eine wichtige Rolle bei der Entdeckung neuer Arzneimittelwirkstoffe und stellen nach wie vor eine Hauptquelle für bioaktive Moleküle für die Arzneimittelentwicklung dar. In jüngster Zeit hat sich die Entdeckung neuer bioaktiver Naturstoffe verlangsamt, da die Bildung der meisten Sekundärmetaboliten, die von biosynthetischen Genclustern kodiert werden, unter Standardkultivierungsbedingungen zum Schweigen gebracht wird und die immense Wiederentdeckung bereits bekannter Produkte im Rahmen der zeit- und arbeitsintensiven traditionellen Screeningverfahren [1].
In den letzten Jahrzehnten wurden verschiedene Methoden wie Ribosomen-Engineering, heterologe Expression, Manipulation oder Regulierung von Promotoren, Induktion von Mutationen, Quorum-Sensing- und Elicitor-Screening-Systeme, Co-Kultivierung und veränderte Kultivierungsbedingungen eingeführt, um stille biosynthetische Gencluster zu aktivieren und die Chance auf die Entdeckung neuer bioaktiver SMs zu erhöhen [2]. Die Manipulation der Kultivierungsbedingungen wurde in der Literatur als der effektivste Ansatz beschrieben, der als OSMAC-Strategie (One strain many compounds) bezeichnet wird. OSMAC beschreibt die Produktion vieler SMs aus einem Mikroorganismus durch die Veränderung der Kulturbedingungen wie Temperatur, pH-Wert, Sauerstoffzufuhr, Kulturgefäß, Medienzusammensetzung, Zusatz von Enzyminhibitoren und kleinen Molekülen [3].
In diesem Projekt sollen durch die Anwendung von Metabolomics-Screening-Strategien neue Sekundärmetaboliten und die Aktivierung von stillen Genclustern in Bakterien mit Hilfe des "One strain many compounds"-Ansatzes entdeckt werden.
[1] Katz, L. and Baltz, R., 2016. Natural product discovery: past, present, and future. Journal of Industrial Microbiology & Biotechnology, 43(2-3), pp.155-176.
[2] Zarins-Tutt, J., Barberi, T., Gao, H., Mearns-Spragg, A., Zhang, L., Newman, D. and Goss, R., 2016. Prospecting for new bacterial metabolites: a glossary of approaches for inducing, activating and upregulating the biosynthesis of bacterial cryptic or silent natural products. Natural Product Reports, 33(1), pp.54-72.
[3] Pan, R., Bai, X., Chen, J., Zhang, H. and Wang, H., 2019. Exploring Structural Diversity of Microbe Secondary Metabolites Using OSMAC Strategy: A Literature Review. Frontiers in Microbiology, 294(10).
Ansprechpartner
Genome Mining und Enzymscreening
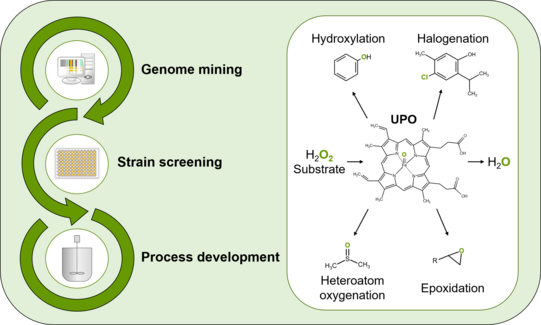
Im Jahr 2004 wurde im Basidiomyceten Agrocybe aegerita eine neue Häm-Thiolat-Peroxidase entdeckt, die von Natur aus ein breites Spektrum an oxidativen Umwandlungen katalysiert [1].
Dieser neuartige Enzymtyp, der als unspezifische Peroxygenasen (UPOs, EC 1.11.2.1) klassifiziert wird, ist im gesamten Pilzreich zu finden und kombiniert den katalytischen Zyklus der Häm-Peroxidasen mit dem "Peroxid-Shunt" der Cytochrom-P450-Monooxygenasen (CYPs). Im Allgemeinen übertragen die promiskuitiven Biokatalysatoren ein Sauerstoffatom von Wasserstoffperoxid auf zahlreiche Substrate und sind daher besonders vielversprechend für Anwendungen, bei denen es um die selektive Oxyfunktionalisierung von organischen Molekülen geht [2].
Aufgrund der ständigen Nachfrage nach neuen Biokatalysatoren mit neuartigen Reaktions- und Substratspektren ist es von großem Interesse, mutmaßliche UPOs zu identifizieren und zu charakterisieren. In diesem Projekt wird eine Kombination aus bioinformatischer Analyse und Enzymaktivitätsscreening eingesetzt, um selten beobachtete Pilzstämme und ihre UPOs als vielversprechende Biokatalysatoren für Hydroxylierungsreaktionen zu identifizieren. Die weitere Optimierung von Wildtyp-Stämmen sowie heterolog exprimierten UPOs kann zur Entwicklung neuer Oxyfunktionalisierungsprozesse für biotechnologische Anwendungen führen.
[1] R. Ullrich, J. Nueske, K. Scheibner, J. Spantzel, and M. Hofrichter, “Novel Haloperoxidase from the Agaric Basidiomycete,” Appl. Environ. Microbiol., vol. 70, no. 8, pp. 4575–4581, 2004
[2] M. Hofrichter, H. Kellner, R. Herzog, A. Karich, C. Liers, K. Scheibner, V. Wambui Kimani, and R. Ullrich, “Fungal Peroxygenases: A Phylogenetically Old Superfamily of Heme Enzymes with Promiscuity for Oxygen Transfer Reactions,” in Grand Challenges in Fungal Biotechnology, P. H. Rampelotto, Springer, pp. 369–403, 2020.
Ansprechpartner
Zyklische Dinukleotide
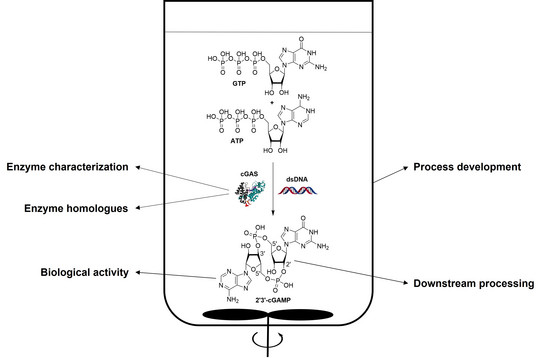
In Anwesenheit von zytosolischer DNA katalysiert das Enzym cyclische GMP-AMP-Synthase (cGAS) die Synthese des zweiten Botenstoffs 2'3'-cyclisches GMP-AMP (cGAMP) [1].
Das zyklische Dinukleotid cGAMP ist derzeit von großem Interesse für die Pharmaindustrie, da es das angeborene Immunsystem aktivieren kann [2]. Mögliche Anwendungen sind die Immuntherapie von Krebs oder die Verwendung als Adjuvans in Impfstoffen [3].
Ziel dieses Promotionsthemas ist die Entwicklung eines enzymatischen Verfahrens zur Herstellung der relevanten zyklischen Dinukleotide.
[1] Sun, L., et al., Cyclic GMP-AMP synthase is a cytosolic DNA sensor that activates the type I interferon pathway. Science (New York, N.Y.), 2013. 339(6121): p. 786-791.
[2] Corrales, L., et al., Direct activation of STING in the tumor microenvironment leads to potent and systemic tumor regression and immunity. Cell reports, 2015. 11(7): p. 1018–1030.
[3] Cai, X., Y.-H. Chiu, and Z.J. Chen, The cGAS-cGAMP-STING pathway of cytosolic DNA sensing and signaling. Molecular cell, 2014. 54(2): p. 289–296.