Zwei neue Publikation sind in Zusammenarbeit mit Prof. Katrin Rosenthal (Constructor University) und Prof. Markus Nett (TU Dortmund) erschienen.
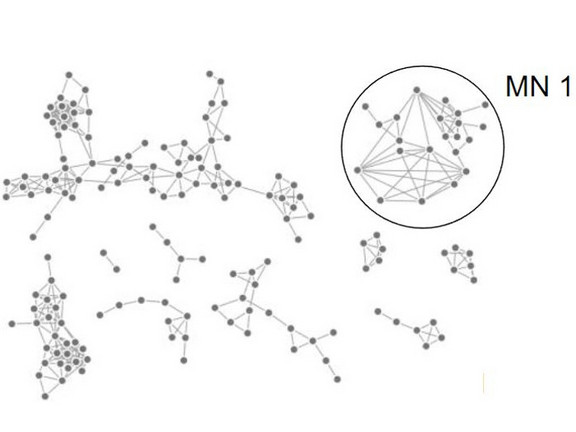
“Molecular Networking-Guided Discovery of Kyonggic Acids in Massilia spp.” in European Journal of Organic Chemistry
Till Steinmetz und seine Mitautoren nutzten Massenspektrometrie basierte molekulare Netzwerke, um die erste umfassende Analyse des Sekundärmetaboloms von Massilia-Arten durchzuführen. Die Arten mit den vielversprechendsten Sekundärmetaboliten wurden mithilfe der molekularen Netzwerke ausgewählt und im großen Maßstab kultiviert. Fünf Metabolite konnten nach Aufreinigung in Milligramm-Mengen gewonnen werden. Die Strukturaufklärung ergab ein zyklisches Dipeptid und vier Acylaminosäuren, die mit der thalassotalic acid C aus dem Meeresbakterium Thalassotalea sp. verwandt sind. Die vier aus Massilia gewonnenen kyonggic acids hemmen das Enzym Tyrosinase. Diese Studie zeigt, wie wichtig Massenspektrometrie-basierte molekulare Netzwerke bei der Entdeckung von neuen bioaktiven Naturstoffen ist.
“Fluent Integration of Laboratory Data into Biocatalytic Process Simulation Using EnzymeML, DWSIM, and Ontologies.” in Processes
Alexander S. Behr und seine Mitautoren entwickeln eine neue Methode für schnelle Screening von Reaktionsparametern in Strömungsreaktoren. Ein neuartige Arbeitsablauf mit digitaler Bildverarbeitung (DIP) wurde entwickelt zur Quantifizierung von Produktkonzentrationen und die Verwendung einer strukturierten Datenerfassung mit EnzymeML-Tabellen in Kombination mit ontologiebasierten semantischen Informationen. Ein wesentlicher Vorteil der Methode ist die schnelle und reibungslose Datenintegration in ein Simulationswerkzeug für die Kinetikbewertung. Eine wichtige Erkenntnis der Studie ist, dass eine flexibel anpassbare Ontologie für die FAIR (Findability, Accessibility, Interoperability, Reusability) Datenverarbeitung unerlässlich ist.